- 只会 Windows 也能轻松搭建远程桌面 RustDesk 自用服务
- JavaWeb开发 —— SpringBootWeb综合案例
- Android 获取网络连接状态新方法
- Linux下搭建安防视频监控系统(NGINX+ffmpeg完成整体项目
- Spring-WebFlux使用,一文带你从0开始学明白Spring-
- 如何开启本地的mysql
- 【框架篇】Spring Boot 日志
- SQL Server 数据库出现可疑标记的解决方法
- 【SpringMVC】JSON注解&全局异常处理机制
- 【Java】Spring MVC程序开发
- failed to open stream: No such file
- 基于java Swing 和 mysql实现的飞机订票系统(源码+数据
- 如何升级npm 和 安装nvm 及 升级node.js
- JAVAWEB学生信息管理系统保姆级教程(增删改查+登录注册+Filt
- Nginx详细教程
- Web项目部署环境搭建:JDK + Tomcat + IDEA +My
- 2023最流行的 Node.js 框架
- QT 5.13 使用MySQL数据库,含MYSQL安装和QT驱动安装及
- 【问题】本地计算机上的MySQL服务启动后停止。某些服务在未有其他服务
- Spring Boot动态设置定时任务
- Spring Boot整合neo4j
- Springboot-Redisson - 1.简介和配置
- RabbitMQ-常用命令
- 记录打包部署Springboot项目到Windows环境
- mac卸载mysql教程(按照步骤可完全卸载)
- Mybatis-plus的自动填充功能
- 【绿色版】Mysql下载、安装、配置与使用(保姆级教程)
- 消息队列之六脉神剑:RabbitMQ、Kafka、ActiveMQ 、
- SpringBoot
- 【SpringBoot快速入门】(4)SpringBoot项目案例代码
生信学习之通路富集一(GO分析):
富集分析的理论知识
富集分析(Enrichment Analysis)是一种广泛应用于生物信息学研究的统计方法,主要用于检验一个基因集合中某些功能或特征的富集程度。富集分析的主要目的是从大量基因数据中找出有生物学意义的模式和功能。根据分析的目标和方法,富集分析可以分为以下几种类型:
基因本体论富集分析(Gene Ontology Enrichment Analysis):这是最常用的富集分析类型,用于检验基因集合中基因本体论(GO)条目的富集情况。这可以帮助研究者了解基因集合中的基因在生物学过程、分子功能和细胞组成方面的共同特征。
通路富集分析(Pathway Enrichment Analysis):这种类型的富集分析主要关注基因在代谢途径和信号传导通路中的作用。通过检测基因集合中通路的富集情况,研究者可以了解这些基因在生物体内的功能和调控机制。通路数据库如KEGG(Kyoto Encyclopedia of Genes and Genomes)和Reactome是进行通路富集分析的常用资源。
基因集富集分析(Gene Set Enrichment Analysis,GSEA):GSEA是一种旨在检测一个基因集合(如差异表达基因)与某些生物学特征(如基因本体论、通路、疾病、表型等)之间的关联的方法。GSEA可以帮助研究者了解基因集合与生物学功能和过程之间的关联,从而揭示潜在的生物学意义。
蛋白质-蛋白质相互作用富集分析(Protein-Protein Interaction Enrichment Analysis):这种类型的富集分析关注蛋白质之间的相互作用,帮助研究者了解基因集合中蛋白质在细胞信号和代谢过程中的功能。
基因表达调控富集分析:这种类型的富集分析关注转录因子、miRNA等调控因子对基因表达的调控作用。通过这种富集分析,研究者可以了解基因表达的调控机制和相互关系。
GO分析的理论知识
基因本体论(Gene Ontology, GO)是一个用于描述基因和基因产品属性的标准术语体系。它提供了一个有组织的方式来表示基因在生物体内的各种角色。基因本体论通常从三个层面对基因进行描述:细胞成分(Cellular Component,CC)、生物学过程(Biological Process,BP)和分子功能(Molecular Function,MF)。
细胞成分(Cellular Component,CC):这个层面描述了基因产物(如蛋白质)在细胞内的定位。例如,它们可能位于细胞核、细胞质、线粒体膜或其他细胞器上。这有助于了解基因产物在细胞内的作用和功能。
生物学过程(Biological Process,BP):这个层面描述了基因参与的生物学过程。这些过程可能包括细胞生长、信号传导、基因表达调控、代谢途径等。通过了解基因参与的生物学过程,我们可以更好地理解生物体的生理功能和疾病发生机制。
分子功能(Molecular Function,MF):这个层面描述了基因产物在分子层面的功能,通常涉及到与其他分子的相互作用或催化生化反应。例如,某个基因产物可能是一个酶,它能够催化某个特定的生化反应;或者它可能是一个结构蛋白,参与细胞骨架的组装和维持。
基因本体论为研究者提供了一个系统的方法来表示和共享关于基因和基因产品在生物体内的功能和过程的知识。这有助于促进基因功能研究的发展,提高研究效率,为疾病治疗和药物研发提供重要信息。
show一些相关的图
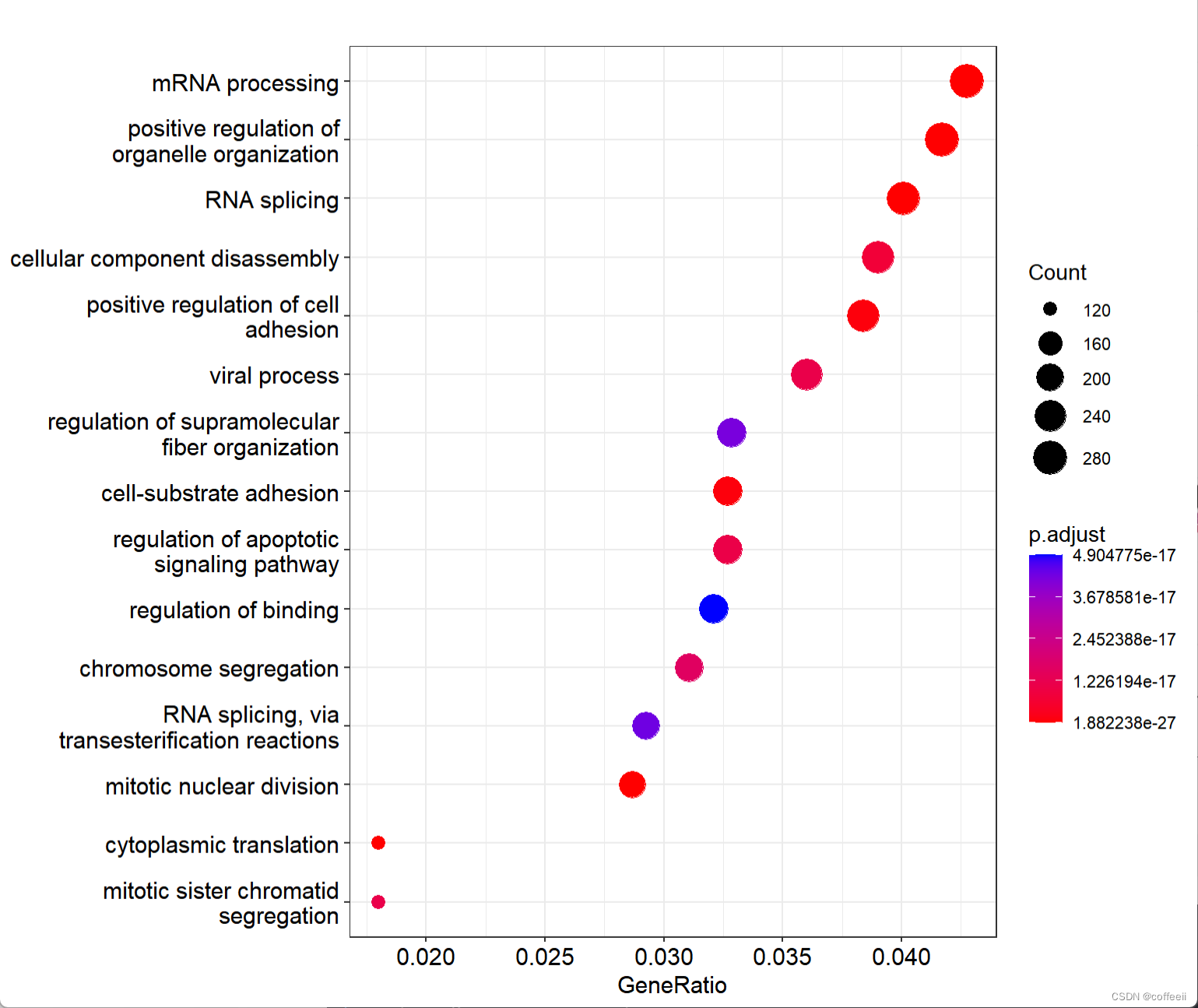
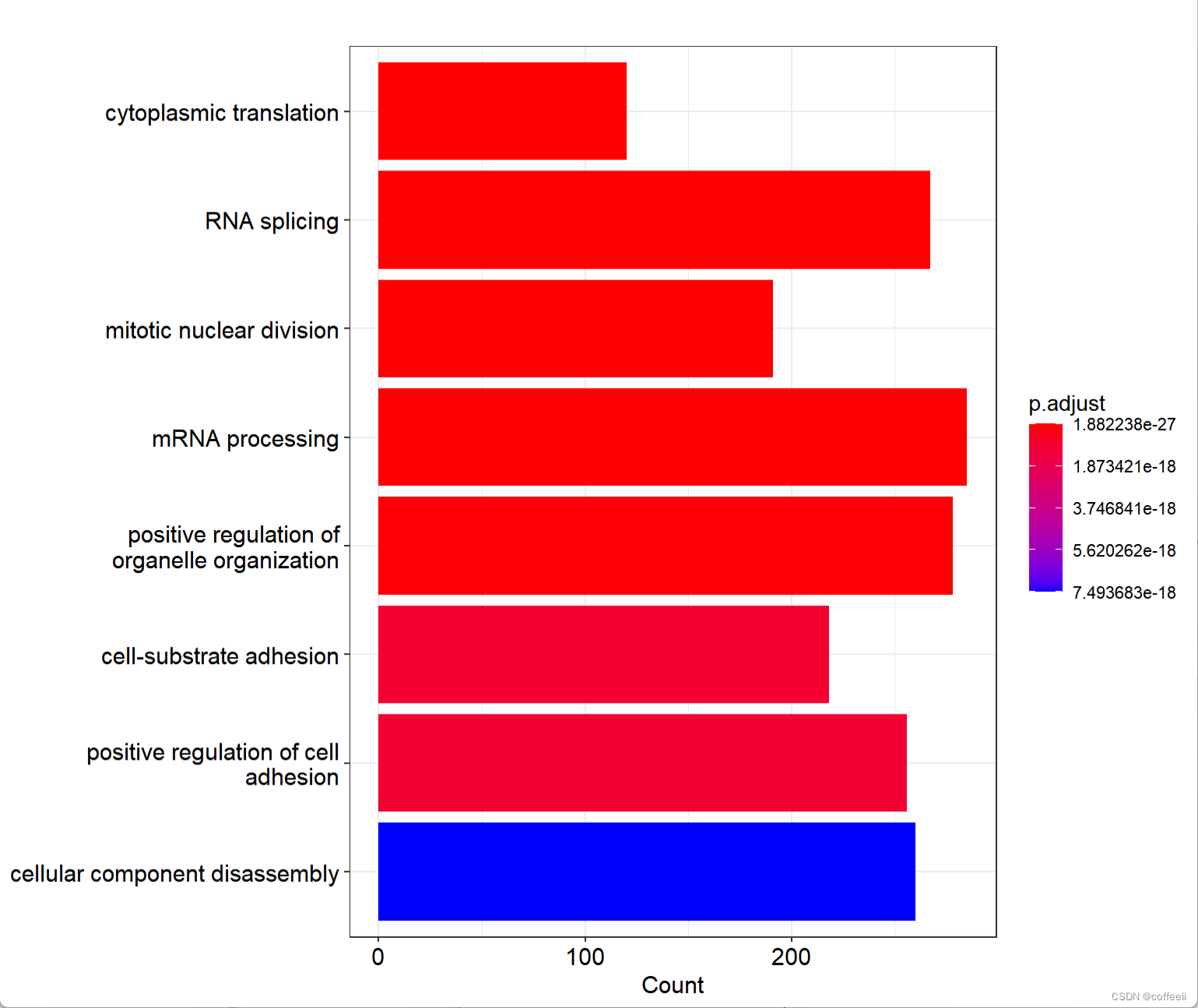
GO分析基本原理:
准备基因列表:首先,你需要一个待分析的基因列表,这通常是从实验数据中获得的差异表达基因或与特定条件相关的基因集合。
映射基因到GO条目:然后,需要将基因列表中的每个基因映射到相应的GO条目。这可以通过使用生物信息学工具和数据库(如DAVID、Ensembl、AmiGO等)来实现。
统计富集情况:接下来,需要计算每个GO条目在基因列表中的富集情况。这通常通过比较实际观察到的基因数目与根据背景基因组随机分布预期的基因数目之间的比例来完成。常用的统计方法包括超几何检验、Fisher精确检验和卡方检验等。
多重比较校正:由于GO富集分析涉及到大量假设检验,因此需要进行多重比较校正,以减少假阳性结果。常用的多重比较校正方法包括Bonferroni校正、Benjamini-Hochberg校正(FDR)等。
解释和可视化结果:最后,可以根据富集分析的结果对基因列表进行解释,找出具有生物学意义的功能、过程和分子功能。此外,还可以通过各种可视化工具(如Cytoscape、REVIGO等)将富集结果以图形形式展示,便于理解和交流。
基因本体论分析通过评估基因集合中特定生物学功能、过程或分子功能的富集程度,帮助研究者从基因表达数据中提取生物学意义。这有助于揭示基因在生物学过程和功能方面的关联,从而促进对生物体内的基因调控和作用机制的理解。
GO分析代码
// An highlighted block
degdf <- FindMarkers(scRNA1,ident.1 = "DapiNeg1",ident.2 = "DapiNeg2",
logfc.threshold = 0.5,group.by = "orig.ident",ident=1)
degdf <- FindAllMarkers(scRNA1)
saveRDS(degdf,"markers.rds")
# degdf<-readRDS("markers.rds")
# BiocManager::install("org.Hs.eg.db")
# BiocManager::install("rlang")
# BiocManager::install("vctrs",force = TRUE)
#
#
#
# install.packages("clusterProfiler")
# install.packages("GOSemSim")
#
#
#
# options(connectionObserver = NULL)
library(org.Hs.eg.db)
library(GOSemSim)
library(clusterProfiler)
degs.list=rownames(degdf)
erich.go.BP = enrichGO(gene =degs.list,
OrgDb = org.Hs.eg.db,
keyType = "SYMBOL",
ont = "BP",
pvalueCutoff = 0.05,
qvalueCutoff = 0.05)
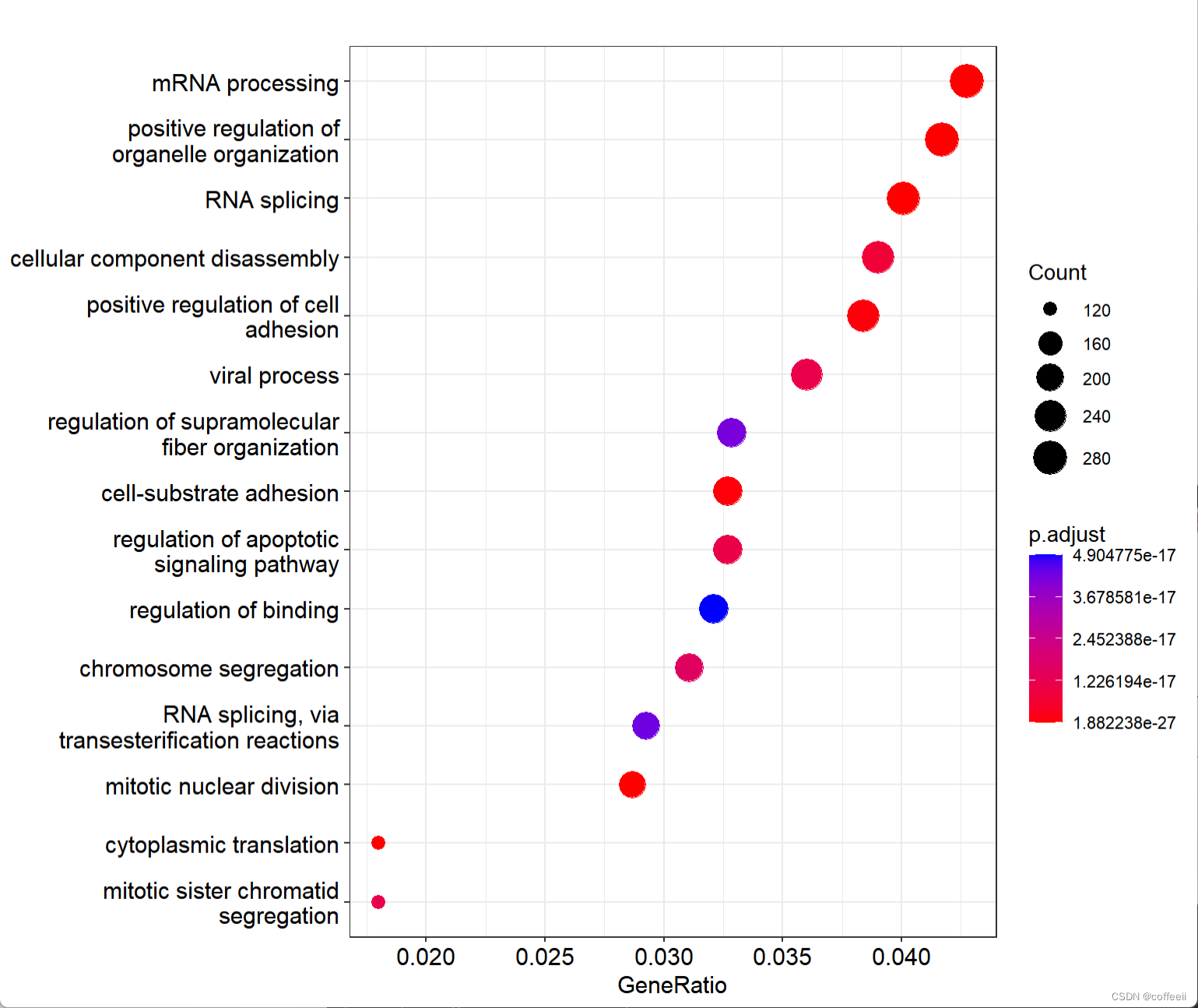
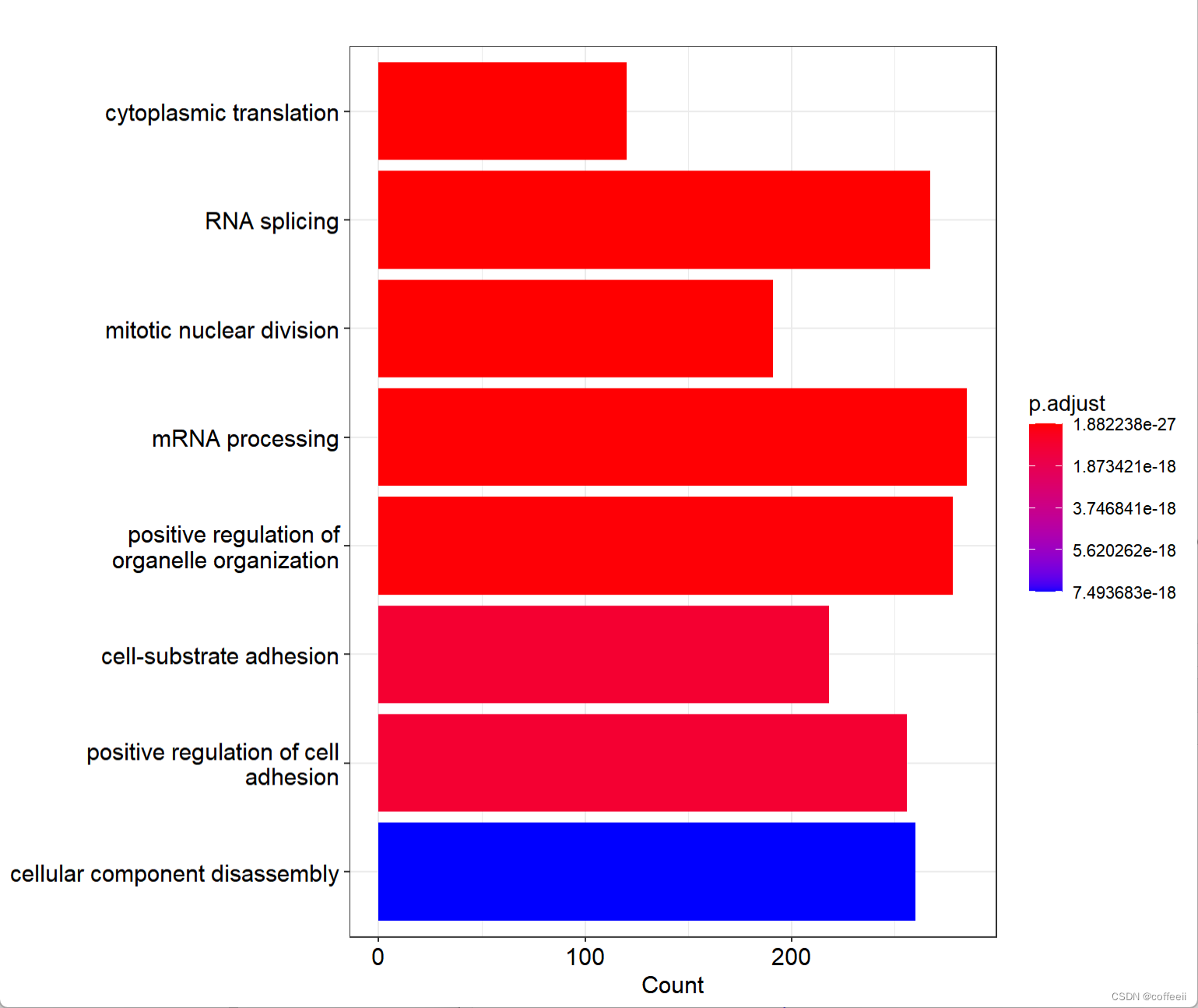
GO项目的创立可以追溯到1998年,其成果和方法已被广泛采用并发表在众多研究论文中。以下是一些关于GO项目的重要出版物:
- Ashburner, M., Ball, C.A., Blake, J.A., Botstein, D., Butler, H., Cherry, J.M., Davis, A.P., Dolinski, K., Dwight, S.S., Eppig, J.T., Harris, M.A., Hill, D.P., Issel-Tarver, L., Kasarskis, A., Lewis, S., Matese, J.C., Richardson, J.E., Ringwald, M., Rubin, G.M., & Sherlock, G. (2000). Gene Ontology: tool for the unification of biology. Nature Genetics, 25(1), 25–29. https://doi.org/10.1038/75556
这篇论文是关于基因本体论项目的创立和初步实现的重要文献。作者介绍了项目背景、目标以及三个主要组成部分(细胞组件、生物过程和分子功能)的概念和实现。
- The Gene Ontology Consortium. (2017). Expansion of the Gene Ontology knowledgebase and resources. Nucleic Acids Research, 45(D1), D331–D338. https://doi.org/10.1093/nar/gkw1108
这篇论文描述了基因本体论知识库和资源的扩展,包括术语、注释和工具的发展。文章还介绍了GO项目在支持基因功能研究方面的最新进展。
更多生信知识欢迎交流v:coffeeiix(也可接单细胞转录组分析培训)














